Motivation
Chemokine oder Chemotaktische Zytokine sind kleine Signalproteine, die weiße Blutzellen (Leukozyten) zur gerichteten Wanderung (Chemotaxis) anregen. Die Wanderung erfolgt entlang eines Chemokin-Gradienten und wird durch die Bindung der Chemokine an G-Protein gekoppelte Rezeptoren und darauffolgende Signalkaskaden induziert. Am Entzündungsort helfen die Leukozyten bei der Eliminierung von Krankheitserregern und bei der Reparatur des Gewebes. Wenn dieser fein abgestimmte Prozess durcheinandergerät können Überreaktionen und entzündliche Erkrankungen die Folge sein. Die Unterdrückung der Erkennung von Chemokinen durch die Leukozyten ist daher von großem Interesse für die Grundlagenforschung und die Therapieentwicklung.
Peptide aus Rezeptorsequenzen
Basierend auf vorhandenen Strukturdaten haben wir in einem Kooperationsprojekt mit der Autonomen Universität Barcelona ein Modell des Chemokins Interlsukin-8 (CXCL8) gebunden an seinen Rezeptor CXCR1 entwickelt. Mit diesem Modell lassen sich die Aminosäuren identifizieren, die für die Protein-Protein-Wechselwirkung von Bedeutung sind. Das Chemokin bindet einerseits an den N-Terminus des Rezeptors (Bindungsstelle 1) und andererseits an die extrazellulären Domänen 3 und 4 (Bindungsstelle 2) wie Abbildung 1 zeigt. In Bindestelle 2 wurden zwei für die Bindung wichtige Peptide identifiziert und verknüpft über einen flexiblen Linker nachsynthetisiert. Das Peptid IL8-RP-Loops bindet mit hoher Affinität an das Chemokin und inhibiert zelluläre Antworten von Leukozyten auf Interleukin-8 wie Rezeptorinternalisierung und Chemotaxis.
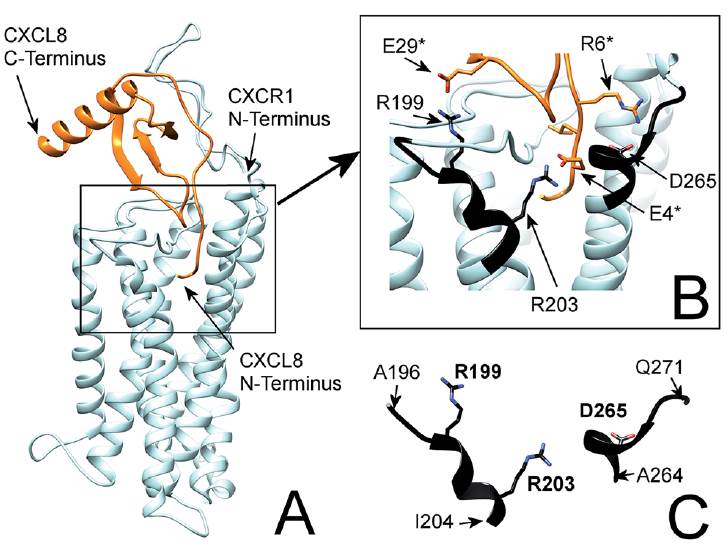
Die Bindepeptide sollen weiter optimiert werden, um höhere Affinität und Stabilität in Serum zu erreichen.
Bindungspartner aus kombinatorischen Peptoid-Bibliotheken
Über kombinatorische Synthese sollen N-substituierte Oligoglycine, sogenannte Peptoide synthetisiert werden, die in der Lage sind an Chemokine zu binden und dadurch die Bindung an ihre jeweiligen Rezeptoren verhindern können.
Dazu wird das Zielchemokin über ein C-terminales Cystein fluoreszent markiert. Mit Hilfe der Mix-and-Split-Methode wird eine Peptoid-Bibliothek mit kurzen Peptoid-Sequenzen am Harz synthetisiert, die mehrere Zehntausend Sequenzen enthält. Im dritten Schritt werden die festphasengebundenen Peptoide mit dem fluoreszenzmarkierten Zielprotein inkubiert. Über die Fluoreszenzmarkierung werden die Peptoide identifiziert, an die das Chemokin bindet. Die Sequenz dieser Peptoide kann massenspektrometrisch ermittelt werden.
Die potenziellen Bindungspartner werden anschließend im größeren Maßstab synthetisiert und auf ihre Bindungsfähigkeit an das entsprechende Zytokin getestet. Auch hier werden vielversprechende Sequenzen chemisch weiter modifiziert, um höhere Affinitäten und biologische Aktivitäten zu erreichen.
Quellen:
Rezeptorpeptid: Helmer, D. et al. RSC Adv., 2015, 5, 25657-25668. Link
Kombinatorische Peptoidbibliotheken: Helmer, D. et al. Anal. Methods, 2016, 8, 4142-4152. Link



